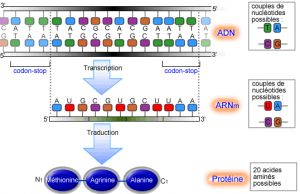
Les bactéries sont des organismes unicellulaires présents dans tous les milieux, y compris les plus hostiles comme les fonds marins ou encore notre intestin. Une bactérie est, en première approximation, une cellule composée d’une chaîne d’ADN baignant dans un milieu visqueux, le cytoplasme, avec d’autres composants comme les ARN, les protéines, etc… Les protéines sont les éléments-clés de la vie cellulaire pour tous les organismes vivants. L’expression maintenant fameuse de F. Crick en 1958, le «dogme central de la biologie moléculaire», affirme que l’information biologique est à sens unique de l’ADN, vers les ARN et ensuite vers les protéines.
À chaque protéine est associée une section de l’ADN, le gène, qui contient le «code» de la protéine. Celle-ci est produite de la façon suivante. Une des macromolécules de la cellule appelées polymérases finit par se fixer après plusieurs essais sur le début du gène pour produire une chaîne de nucléotides, l’ARN messager qui est porteur de la copie du code de la protéine. Puis elle se détache pour évoluer dans le cytoplasme. C’est l’étape de la transcription. Après cet instant d’autres macromolécules, les ribosomes en particulier, peuvent se fixer sur l’ARN messager pour produire une chaîne d’acides aminés (polypeptides) correspondant à la protéine. C’est l’étape de la traduction.
Le cytoplasme de la bactérie est un milieu désordonné dans lequel se déplacent par diffusion les différents composants de la production de protéines: polymérases, ARN, ribosomes, protéines, … Les instants de rencontre polymérase-gène ou ribosome-ARN ne peuvent être complètement contrôlés en raison des fluctuations du milieu, de l’agitation thermique perturbant les liaisons, etc. Ainsi, ce processus comporte une composante aléatoire très importante. Une bactérie comme Escherichia coli compte quelques millions de protéines, de 2000 types différents et de concentrations variant de quelques copies à une centaine de milliers. Les protéines représentent environ la moitié du poids de la bactérie (en ne comptant pas le poids de l’eau) et leur production absorbe un peu plus de 80% de l’ensemble des ressources nécessaires à la croissance des bactéries. En raison du coût de leur production, une des questions-clés de ce domaine est de comprendre les mécanismes cellulaires qui permettent de minimiser la variance (i.e., la variabilité autour de sa valeur moyenne) du nombre de protéines de chaque type.
Les premiers modèles mathématiques de production de protéines ont été développés à la fin des années 1970, bien avant qu’on puisse observer les fluctuations de la concentration des protéines à l’échelle d’une unique bactérie. La modélisation mathématique a néanmoins permis de souligner les conséquences attendues du caractère stochastique du mécanisme de production des protéines. Le cadre mathématique est celui des processus de Markov dans lequel ont été obtenus des résultats concernant la représentation de la variance du nombre de protéines d’un type fixé. Ces premiers résultats ont permis de comprendre un peu mieux le rôle de plusieurs paramètres biologiques de la cellule dans la variabilité de la production des protéines, en particulier de souligner l’existence d’une dépendance directe entre la variance et la moyenne. Dans la deuxième moitié des années 1990, le développement de méthodes efficaces de comptage de protéines, et spécifiquement l’introduction d’une protéine fluorescente appelée GFP (Green fluorescent protein) et depuis d’autres molécules fluorescentes «rapportrices», a conduit à la constitution d’ensembles de données réelles conséquents et appelant leur comparaison avec des modèles mathématiques.
Il n’en reste pas moins que cette connaissance quantitative et les modèles mathématiques explorés sont encore à l’heure actuelle très insuffisants. En particulier, l’importance des différents mécanismes de régulation de la production est encore mal connue, de nouveaux modèles mathématiques doivent être développés et analysés, validés avec des données réelles pour améliorer la compréhension de ce processus biologique fondamental qui se trouve présent au cœur de nos propres cellules.
Brève rédigée par Philippe Robert (INRIA et École Polytechnique) d’après ses travaux avec Vincent Fromion et Emanuele Leoncini (INRA).
Pour en savoir plus :
- J. Paulsson, Models of stochastic gene expression, Physics of Life Reviews 2 (2005), no. 2, 157-175.
- D. Rigney, Stochastic model of constitutive protein levels in growing and dividing bacterial cells, Journal of Theoretical Biology 76 (1979), no. 4, 453-480.
- Y Taniguchi, P.J. Choi, ,G.W. Li, H. Chen, M. Babu, J. Hearn, et Xie, X. S. (2010). Quantifying E. coli proteomeand transcriptome with single-molecule sensitivity in single cells. Science, 329(5991), 533-538.
Crédits images : Wikipedia Commons.