Les propagations de maladies infectieuses, chez l’humain, l’animal et le végétal, peuvent avoir des effets considérables sur une société, quel que soit son stade de développement, jusqu’à mettre en péril aujourd’hui certaines espèces de végétaux et d’animaux. Pour améliorer notre capacité à prévenir et contenir les propagations de maladies infectieuses, nous avons besoin de comprendre comment les pathogènes, c’est-à-dire les virus, les bactéries, ou les champignons par exemple, se disséminent dans les populations d’hôtes. Ainsi, savoir « qui a infecté qui » au cours d’une épidémie est une information essentielle, mais souvent non disponible et difficile à reconstruire. Le travail même de modélisation de l’épidémie est compliqué (voir Les difficultés de la modélisation mathématique des épidémies). Il est toutefois possible de recourir à des indices tels que le temps, l’espace et la génétique et d’en déduire, à l’aide de la statistique notamment, les évènements de transmissions de la maladie.
Au cours d’une épidémie, les hôtes infectés qui sont proches dans le temps et l’espace sont souvent supposés comme étant reliés. Ces deux indices permettent donc d’identifier un ensemble probable de scénarios de transmission de la maladie. Dans l’enquête sur « qui a infecté qui », la génétique peut fournir un indice supplémentaire pour les pathogènes dont le génome évolue rapidement (par exemple des virus à ARN). En effet, même pour des épidémies courtes, on peut identifier des hôtes qui sont porteurs de variants du pathogène plus ou moins proches génétiquement. Ainsi, en plus de la proximité dans le temps et l’espace des hôtes infectés, la proximité génétique des variants pathogènes peut être utilisée pour réduire le nombre de scénarios de transmissions possibles et donc mieux comprendre les déterminants d’une épidémie.
La modélisation bayésienne hiérarchique en combinant de manière quantitative les informations temporelles, spatiales et génétiques relevées au cours d’une épidémie, permet de lever en partie le voile sur « qui a infecté qui ».
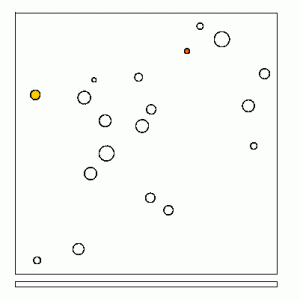
Sur l’image ci-dessus, les hôtes sont représentés par les disques (vides ou pleins). Les particules émises par les hôtes infectieux et servant à la dissémination de la maladie sont représentées par les points noirs. Lorsqu’un point noir tombe dans un disque, l’hôte correspondant à ce disque est infecté. La taille d’un disque est proportionnelle à la réceptivité de l’hôte associé. Un disque non coloré correspond à un hôte non infecté. Un disque coloré correspond à un hôte infecté et infectieux. La couleur du disque reflète le pouvoir infectieux de l’hôte (rouge : très infectieux ; jaune : peu infectieux).
Brève rédigée par Samuel Soubeyrand (INRA) d’après les travaux de Marco J. Morelli, Gaël Thébaud, Joël Chadœuf, Donald P. King, Daniel T. Haydon et Samuel Soubeyrand de l’Université de Glasgow, de l’INRA et de l’Institut pour la Santé Animale de Pirbright.
Pour en savoir plus :
- M.J. Morelli, G. Thébaud, J. Chadœuf, D.P. King, D.T. Haydon, S.Soubeyrand (2012), «A bayesian inference framework to reconstruct transmission trees using epidemiological and genetic data», PLOS Computational Bilogy, Vol.8, No. 11 [en anglais].
- Brève connexe : Les difficultés de la modélisation mathématique des épidémies.
- Un article sur Interstices pour comprendre comment modéliser la propagation d’une épidémie.
Crédits Images :
- Photo Colin Smith
- Simulation Samuel Soubeyrand




Je vous remercie pour le raisonnement et l’explication assez claire sur la statistique enquête sur le temps, l’espace et la génetique.